16S full length rDNA Sequencing 16S full length rDNA Sequencing
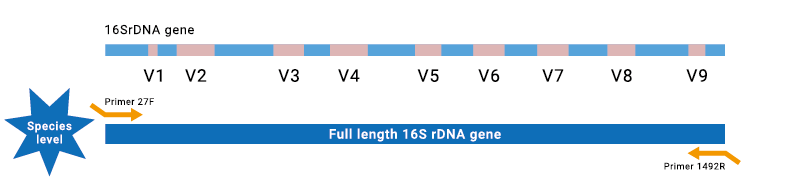
16S rDNA 是細菌中的一種保守基因,長度約為 1500nt,DNA 序列包含 V1~V9 高度變異的區域,即使在相近的物種之間也有差異的特性,使得 16S 區域的序列分析在細菌分類學或鑑定中被廣泛使用。我們利用 PCR 引子擴增全長 16S rDNA,然後透過 PacBio HiFi 長讀長的定序技術,提供高精確性的定序結果,達到 "物種" 級別分類和鑑定。
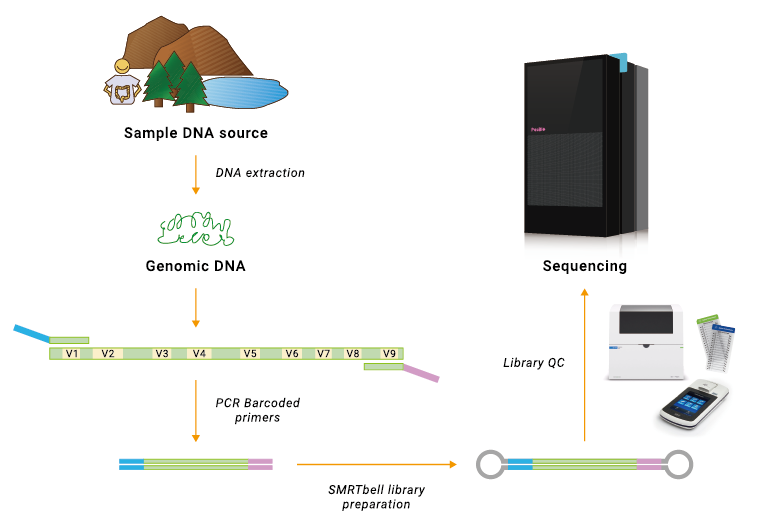
自樣品萃取之細菌 DNA,利用 1-Step PCR 擴增全長 16S rDNA (V1-V9) 區域的片段,同時於 5‘ 端連接特定的 Barcode 序列。隨後,將雙股 PCR 產物接上 PacBio 特殊設計的髮夾彎序列,製備成啞鈴狀模板 (SMRTbell) 文庫,並於 PacBio Revio Platform 進行定序。
樣品條件
| DNA QC 標準 | ||
|---|---|---|
| 樣品種類 | 純菌 DNA | 含 host DNA |
| 總量 (Qubit, dsDNA) | ≥150 ng | ≥300 ng |
| 體積 | ≥15 µL | ≥15 µL |
| 濃度 | ≥10 ng/uL | ≥20 ng/uL |
| 品質 (TapeStation) | major peak ≥ 10kb | major peak ≥ 10kb |
定序策略
- PacBio Revio Platform
- 每樣品 10,000 HiFi Reads
分析流程
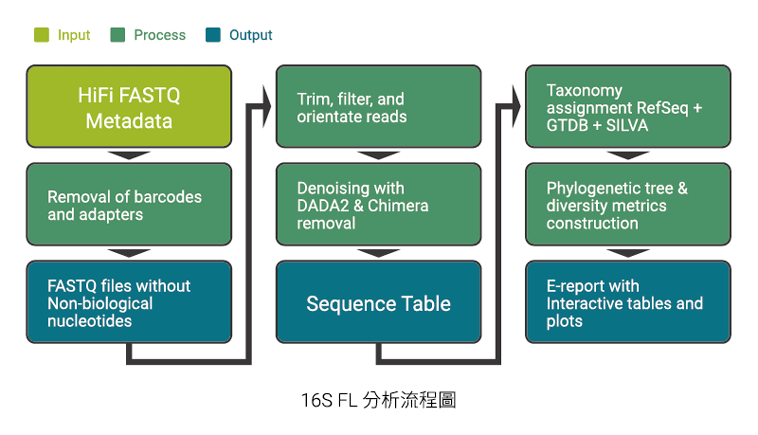
PacBio SMRTLink 被用於校正定序下機的原始數據並生成 CSS reads。Q 值達到 Q20 的 CCS reads 被定義為 HiFi reads。樣品拆分後,我們去除了序列中的樣品標籤和非生物鹼基的接合子 (adapter)。接著,我們根據序列的錯誤率和長度篩選掉低品質序列,並將保留之序列它們校正為一致的方向,然後運行 DADA2 演算法降噪,以獲得 ASVs (Amplicon Sequence Variants) 的特徵序列。
我們使用了三個資料庫,包括 NCBI 16S ribosomal RNA sequences (BioProject 33175 和 33317 的 RefSeq 記錄),Genome Taxonomy Database (GTDB) 和 SILVA database20,並使用 Naive Bayesian Classifier 對這些 ASVs 進行物種註釋。此外,我們還利用親緣關係樹估算組內 (alpha) 和組間 (beta) 的多樣性,結合不同統計模型的結果和互動式圖表,生成網頁報告。
16S 全長標準分析 - Diversity analysis
- 分析結果為向量檔案格式,不失真且容易編輯。
- 提供敘述性統計和整體與胼對檢定結果,透過互動式表格可快速篩選出符合條件之結果。
Rarefaction curve
Observed features 和 Shannon's index 之運算結果
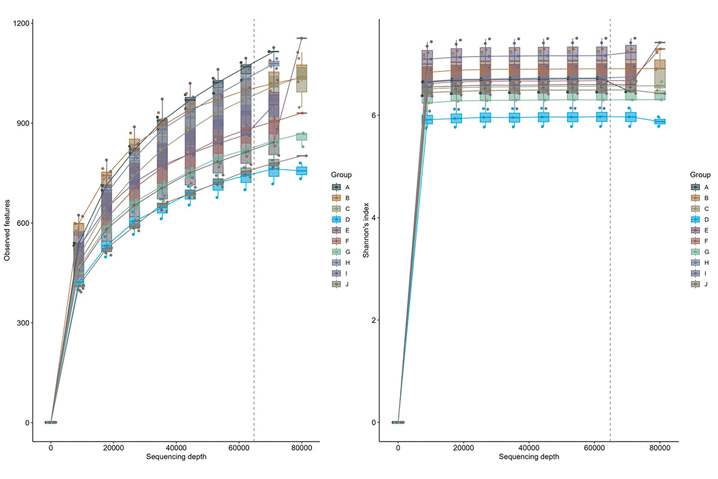
α diversity
7 種多樣性指數計算

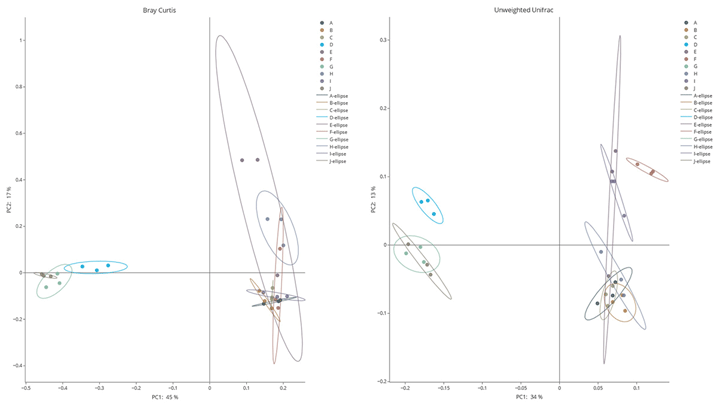
β diversity
6 種多樣性指數計算
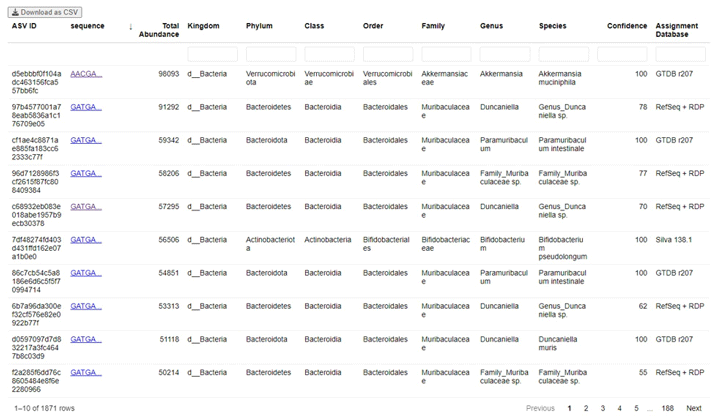
16S 全長標準分析- Taxonomic assignment
- 提供 ASV 之物種註釋結果,和樣品豐度資訊。
- 以各式互動圖表分析樣品物種組成。
Representative sequence and taxonomy assignment per ASV
每個 ASV 的總量和對應之物種註釋,點選序列即可進行線上 BLAST。
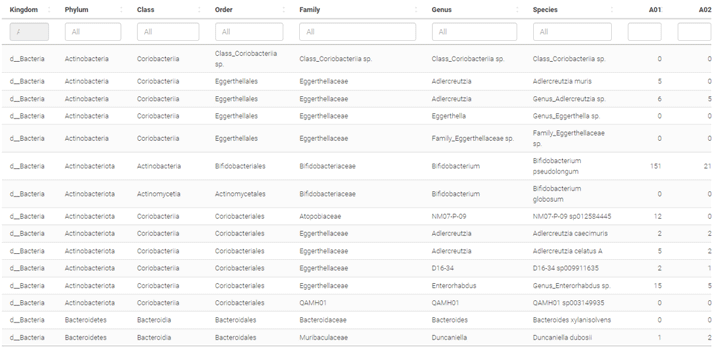
Taxonomy abundance table
提供絕對豐度和相對豐度之結果表格
Barplot of taxonomic classification
互動式介面調整樣品排序和整體配色
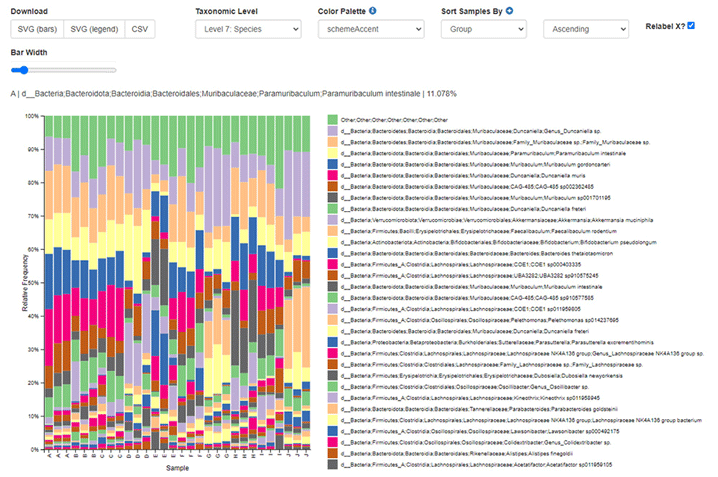
16S 全長標準分析 - 物種組成
- 提供 ASV 之物種註釋結果,和樣品豐度資訊。
- 以各式互動圖表分析樣品物種組成。
Krona Interactive Pie Chart
以圓餅圖顯示樣品之物種組成

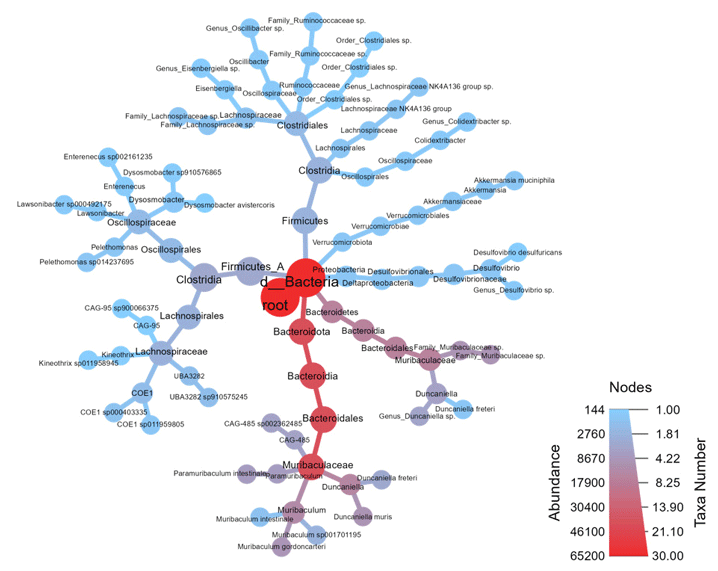
Heat Tree Plot
查看各分類階層之物種組成
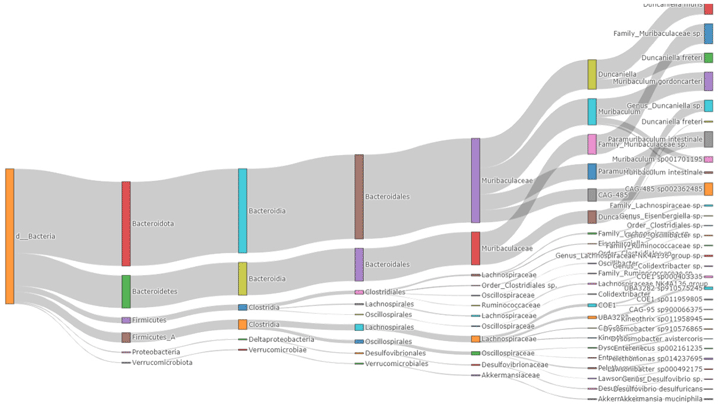
Sankey plot
展示樣品菌種在各分類階層之變化
16S 全長標準分析 - 群組分析
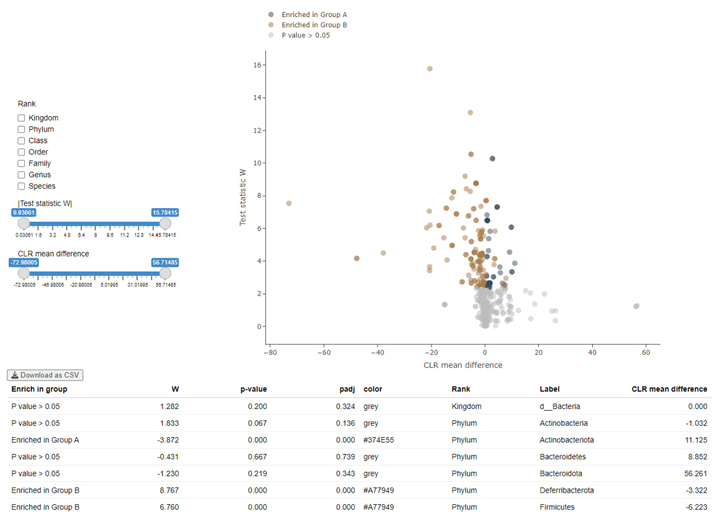
Differential abundance analysis- 對不同組別差異性顯著影響之物種。
ANCOM-BC
(Analysis of Compositions of Microbiomes with Bias Correction)
駢對檢定,分析結果以火山圖顯示差異菌種,可以互動式介面調整參數。
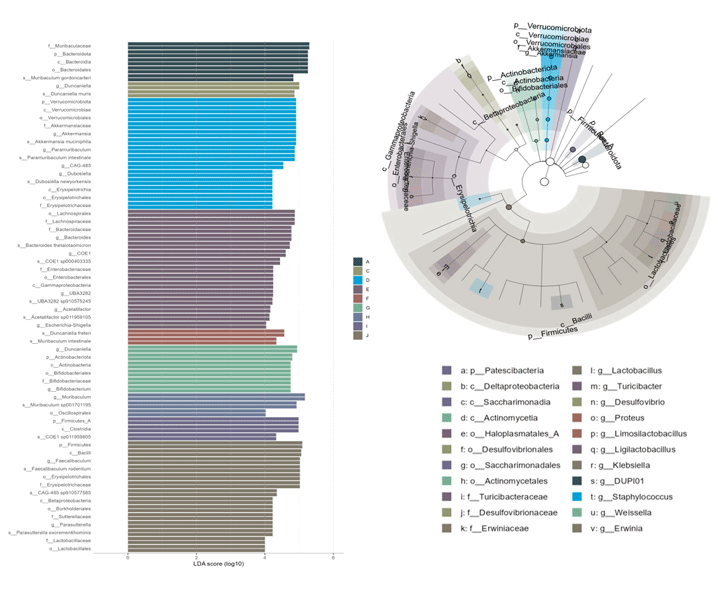
LEfSe (Linear discriminant analysis Effect Size)
進行多組比較,分析結果以直條圖和分支圖呈現。
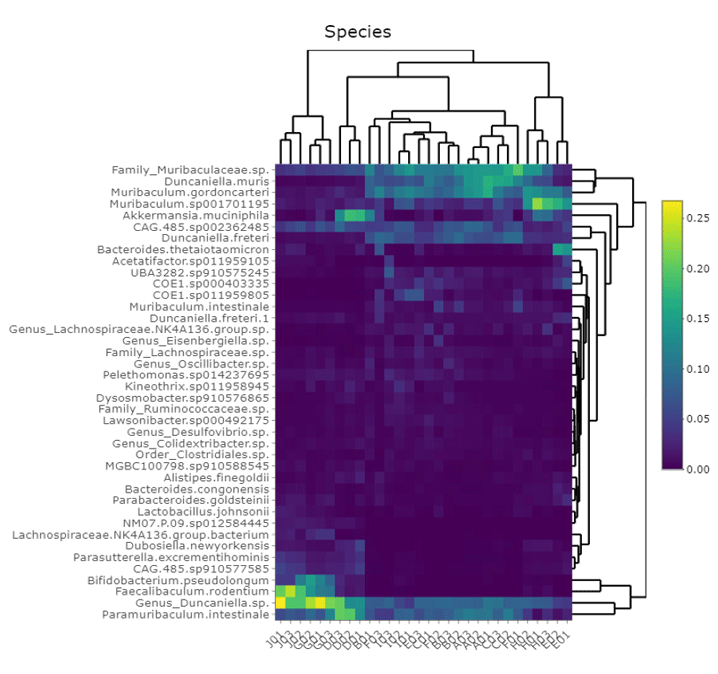
Hierarchical Clustering Heatmap with Dendrograms
以物種之豐度進行樣品聚類熱圖繪製
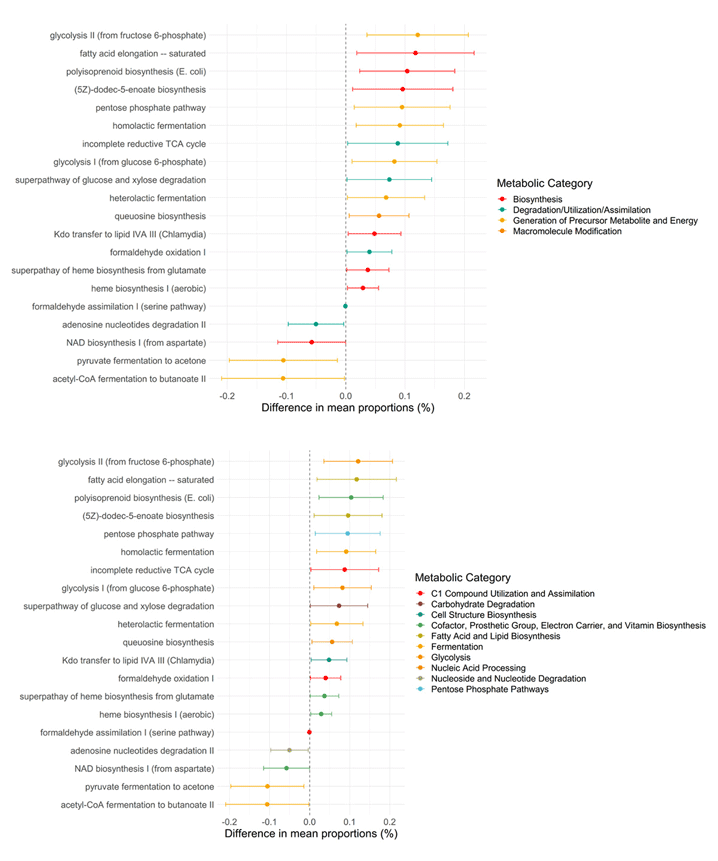
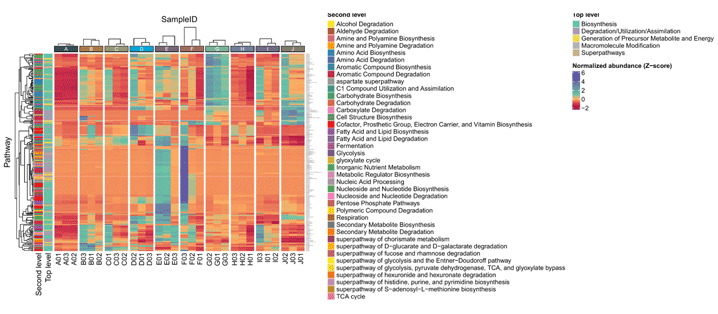
16S 全長標準分析 - 細菌功能性預測分析
Functional analysis- 以 PICRUSt2 (Phylogenetic Investigation of Communities by Reconstruction of Observed States) 進行功能性預測。
- 使用 MetaCyC 和 KEGG 資料庫。


- 提供敘述性統計和整體與胼對檢定結果,透過互動式表格可快速篩選出符合條件之結果。
Heatmap
顯示多組比較結果
Forest plot
顯示駢對比較結果